工学院大学 生体生命情報研究室 [日本語] [English]
次世代シーケンサのデータ処理
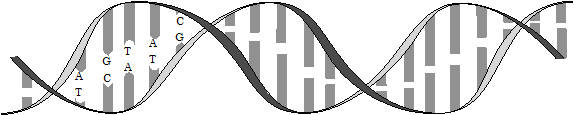
生物の遺伝情報は親から子へと引き継がれていきます。この遺伝情報はDNA(デオキシリボ核酸)という分子に格納されています(下図参照)。DNAは糖(デオキシリボース)、リン酸、塩基の3つからなるヌクレオチドというものが多数結合してできています。塩基にはアデニン(A)、チミン(T)、グアニン(G)、シトシン(C)の4種類があります。生物の遺伝情報の実体は、このATGCの塩基の並びです。塩基の並び方によって、タンパク質の設計図である遺伝子がコードされています。
設計図である遺伝子の塩基配列が通常と異なっている(配列変異)と、異常なタンパク質ができる可能性があります。したがって、遺伝子の塩基配列を調べることによって、疾患にかかる可能性がどの程度あるかを予測できると考えられています。また、がん細胞では配列変異が生じていることが多々あり、これが抗癌剤に対する耐性などに関係していると考えられています。
このような背景から、DNAの塩基配列を高速に調べる方法が開発されています。そのような方法を次世代シーケンサ(NGS)と呼びます。ヒトのDNAは約30億もの塩基が並んだものです。読取りの精度を高めるために何度か読むことを考えると、NGSのデータはしばしばテラバイトオーダーの膨大な量になります。下図はヒトのDNA配列の一部を示したものです。ATGCの4種類の文字がたくさん並んでおり、目で見てもその意味はわかりません。そこでコンピュータを使って解析することになります。
NGSのデータには個人の変異とNGSによる読み間違えが混在しています。現状では膨大なデータの処理に非常に時間がかかっており、疾患の可能性や抗癌剤の耐性に関連する配列変異などの有益な情報を高速に抽出する方法の開発が望まれています。
そこで本研究室では、NGSのデータ処理についてさまざまな角度から研究を行っています。1つは有益な情報を抽出するために多種のデータと統合的に解析する方法の開発です。また、GPGPUを用いて処理を高速化する方法についても研究しています。
なお、この研究テーマは東京医科大学 医学総合研究所 分子腫瘍研究部門との共同研究です。
連絡先
工学院大学工学部
電気電子工学科※
生体生命情報研究室
〒163-8677 新宿区西新宿1-24-2
wwc1059[at]www.ns.kogakuin.ac.jp
*[at] は @ に置換えてください。
※2017年度より電気システム工学科から名称変更
プライバシーについて
本サイトではよりよい情報提供のためにGoogle アナリティクスを使用しています。データ収集の際、cookieを設定したり、既存のcookieを読み取る場合があります。収集する情報の範囲は目的達成に必要な限度を超えないものとします。

